Replicación del ADN
¿Qué es el ADN?
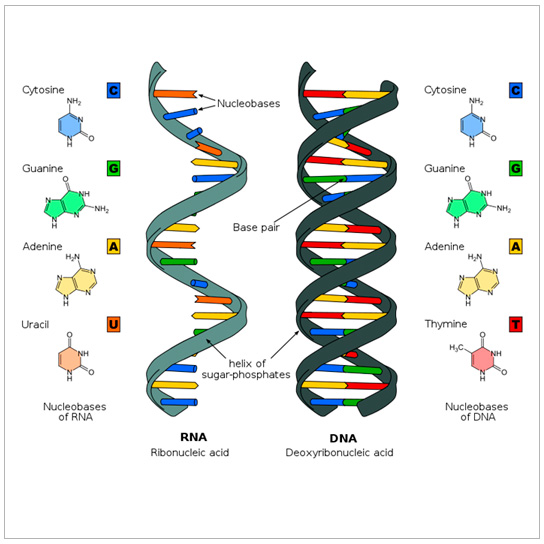
Es el nombre químico de la molécula que contiene la información genética en todos los seres vivos.
Consiste:
De dos cadenas que se enrollan entre ellas para formar una estructura de doble hélice.
Cada cadena tiene una parte central formada por azúcares (desoxirribosa) y grupos fosfato.
Enganchado a cada azúcar hay una de las siguientes 4 bases: adenina (A), citosina (C), guanina (G), y timina (T).
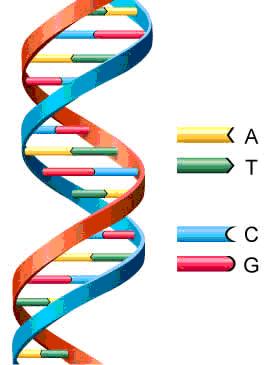
Las dos cadenas se mantienen unidas por enlaces entre las bases; la adenina se enlaza con la timina, y la citosina con la guanina.
La secuencia de estas bases a lo largo de la cadena es lo que codifica las instrucciones para formar proteínas y moléculas de ARN.
¿Qué es la replicación del ADN?
Es el mecanismo que permite al ADN duplicarse (es decir, sintetizar una copia idéntica). De esta manera, de una molécula de ADN única, se obtienen dos o más "réplicas" de la primera y la última.

Se produce:
Esta duplicación del material genético se produce de acuerdo con un mecanismo semiconservador, lo que indica que los dos polímeros complementarios del ADN original, al separarse, sirven de molde cada una para la síntesis de una nueva cadena complementaria de la cadena molde, de forma que cada nueva doble hélice contiene una de las cadenas del ADN original.
Características
Una de las características más notables del ADN es su capacidad de replicarse; dicho de otra manera, tiene la capacidad de formar copias de sí mismo.
La síntesis de las cadenas de ADN durante la replicación se lleva a cabo en dirección 5’ → 3’ tanto en eucariotas como en procariotas. Solamente el carbono de la posición 3’ de la pentosa posee un radical hidroxilo (OH) libre, con el que puede formar un nuevo enlace fosfodiéster con otro desoxirribonucleótido y formar así la hebra creciente de ADN; por esta razón, la cadena de ADN sólo puede crecer en dirección 3’. A este proceso se le llama polimerización, que consiste en la unión de un dNTP (desoxirribonucleótido) complementario a la hebra molde según la Ley de Chargaff.
La replicación del ADN cuenta con tres características que la definen y permiten entender el proceso: semiconservadora, bidireccional y antiparalela.
Importancia
El proceso de replicación tiene su importancia en producir copias de sí mismo por medio de enzimas, pues esto va a originar moléculas de ADN, siendo un proceso semiconservativo porque cada uno de los dos ADN hijos tiene una cadena del ADN anterior y otra nueva que contienen toda la información biológica del organismo.
Puntos importantes
La replicación del ADN es semiconservativa. Cada cadena de la doble hélice funciona como molde para la síntesis de una nueva cadena complementaria. Este proceso nos lleva de una molécula de inicio a dos moléculas "hijas", en las que cada nueva doble hélice contiene una cadena nueva y una vieja.
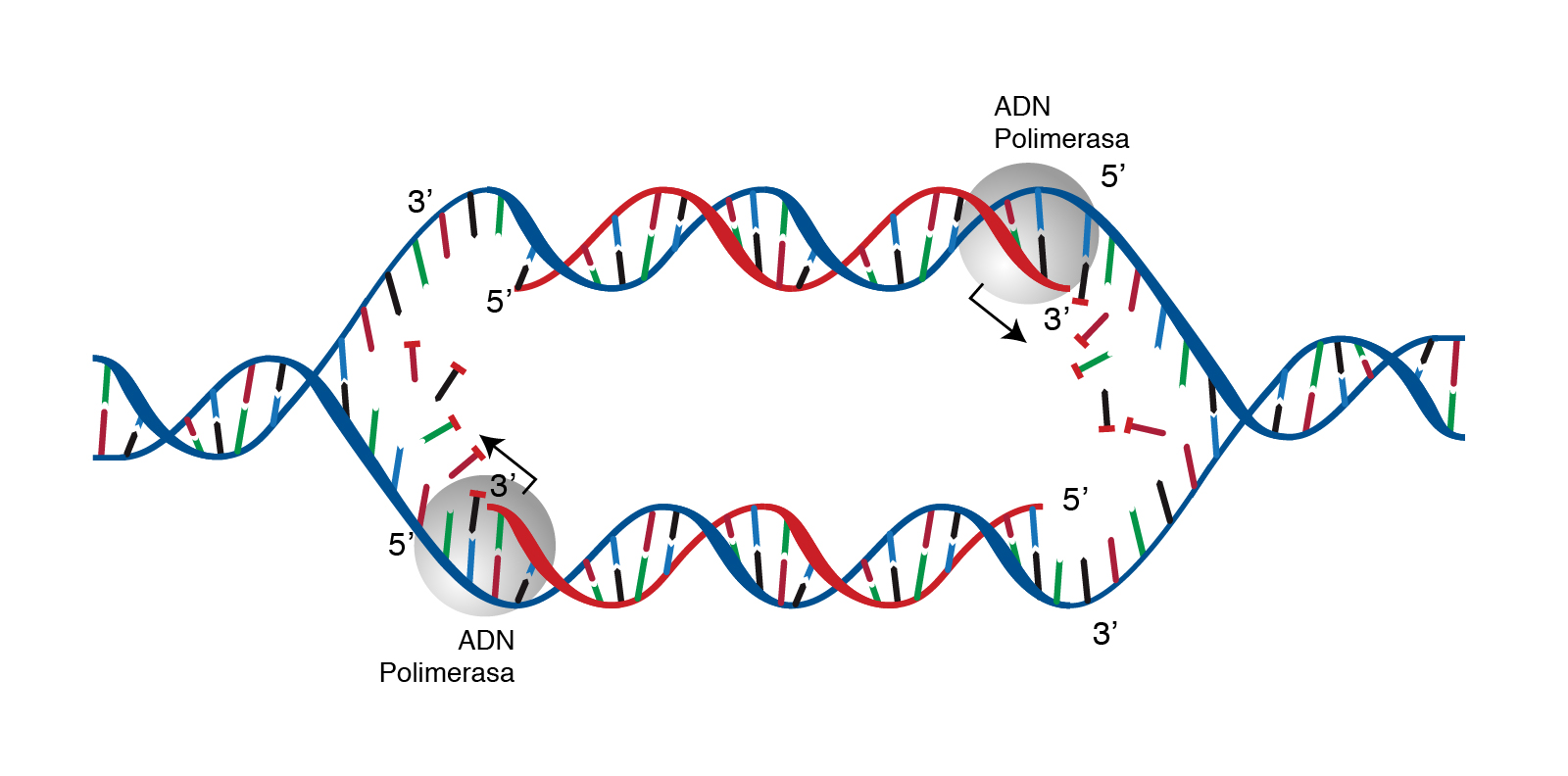
Enzimas llamadas ADN polimerasas producen el ADN nuevo, estas requieren de un molde y de un cebador (iniciador), y sintetizan ADN en dirección 5' a 3'.
Durante la replicación del ADN, una de las cadenas nuevas (la cadena líder) se produce como un fragmento continuo. La otra (la cadena rezagada) se hace en pequeños fragmentos.

La replicación requiere de otras enzimas además de ADN polimerasa, como el ADN primasa, el ADN helicasa, el ADN ligasa y la topoisomerasa.
Lo más interesante de este proceso es cómo lo realiza una célula.
Las células necesitan copiar su ADN muy rápidamente y con muy pocos errores (o se arriesgan a problemas como el cáncer). Para ello, utilizan una variedad de enzimas y proteínas que trabajan en conjunto para asegurar que la replicación del ADN se lleva a cabo sin incidentes y con precisión.
La helicasa desenrolla la hélice y las proteínas de unión a cadenas sencillas impiden que se vuelva a formar. La helicasa abre el ADN en la horquilla de replicación. Las proteínas de unión a cadenas sencillas cubren el ADN alrededor de la horquilla de replicación para evitar que el ADN se vuelva a enrollar.
La topoisomerasa impide que el ADN se enrolle demasiado por delante de la horquilla de replicación. En este proceso, la topoisomerasa puede aliviar la tensión de torsión de la estructura tanto en superenrollamientos negativos como positivos.
El ADN primasa forma un cebador de ARN.
El ADN polimerasa, Son responsables de la síntesis de ADN, añaden nucleótidos uno por uno a la cadena creciente de ADN, e incorporan solo aquellos que sean complementarios al molde. Se extiende la cadena de ADN a partir del cebador de ARN. La síntesis de ADN solo ocurre en dirección 5' a 3'. En la cadena líder, la síntesis de ADN ocurre continuamente. En la cadena rezagada, la síntesis de ADN reinicia muchas veces conforme se desenrolla la hélice, lo que produce muchos fragmentos pequeños llamados "fragmentos de Okazaki". ". Los cebadores de ARN se eliminan y el ADN polimerasa los sustituyen por ADN.
Estas son algunas características clave del ADN polimerasas:
Siempre necesitan un molde.
Solo pueden agregar nucleótidos al extremo 3' de la cadena de ADN.
No pueden comenzar una cadena de ADN desde cero, sino que requieren de una cadena preexistente o segmento corto de nucleótidos llamado cebador.
Pueden corregir, o revisar su trabajo, eliminando la gran mayoría de nucleótidos "equivocados" que se agregan accidentalmente a la cadena.
La adición de nucleótidos requiere energía. Esta energía proviene de los nucleótidos mismos, que tienen tres fosfatos unidos a ellos (muy similar a la molécula portadora de energía ATP). Cuando se rompe el enlace entre los fosfatos, la energía liberada se utiliza para formar un enlace entre el nucleótido entrante y la cadena creciente.
El ADN ligasa une los fragmentos de Okazaki en una sola molécula de ADN.
Bibliografía
Replicación de ADN - Wikipedia, la enciclopedia libre
Replicación | Biología molecular. Fundamentos y aplicaciones ...
Mecanismos moleculares de la replicación del ADN (artículo ...
LA REPLICACIÓN
ADN (Ácido Desoxirribonucleico) | NHGRI
El ciclo celular | CancerQuest
Topoisomerasa II – ChemEvol – UAH
Qué importancia tiene el proceso de replicación - Brainly.lat
¿Cómo ocurre?
La replicación siempre comienza en lugares específicos del ADN, que se llaman orígenes de replicación y se reconocen por su secuencia.
Proteínas especializadas reconocen el origen, se unen a este sitio y abren el ADN. Conforme se abre el ADN, se forman dos estructuras en forma de Y llamadas horquillas de replicación, en conjunto conforman lo que se llama burbuja de replicación. Las horquillas de replicación se mueven en direcciones opuestas a medida que avanza la replicación. En cada horquilla de replicación se sintetiza nuevo ADN complementario a cada cadena.
La helicasa es la primera enzima de la replicación que se carga en el origen de replicación. El trabajo de la helicasa es permitir el avance de las horquillas de replicación "desenrollando" el ADN (rompiendo los puentes de hidrógeno entre los pares de bases nitrogenadas).
Proteínas llamadas proteínas de unión a cadenas sencillas, cubren las cadenas de ADN separadas cerca de la horquilla de replicación, impidiéndoles volver a unirse en una doble hélice.
La primasa hace un cebador de ARN, un corto segmento de ácido nucleico complementario al molde, que proporciona un extremo 3' con el que el ADN polimerasa puede trabajar. Un cebador típico es de cinco a diez nucleótidos de largo. El cebador ceba el ADN polimerasa, es decir, le proporciona lo que necesita para funcionar. Una vez que el cebador de ARN está en su sitio, el ADN polimerasa lo "extiende", añadiendo nucleótidos uno a uno para hacer una cadena nueva de ADN complementaria a la cadena molde.
El ADN polimerasas solo pueden agregar nucleótidos en el extremo 3' de una cadena de ADN existente, ya que utilizan el grupo -OH libre en el extremo 3' como un "gancho" y añaden un nucleótido a este grupo en la reacción de polimerización. El ADN polimerasa que se encarga de la mayor parte de la síntesis El ADN polimerasas solo pueden hacer ADN en dirección 5' a 3', esto plantea un problema durante la replicación. Una doble hélice de ADN siempre es antiparalela; en otras palabras, una cadena corre en dirección 5' a 3', mientras que la otra corre de 3' a 5'. Esto hace necesario que las dos cadenas nuevas, que también son antiparalelas a sus moldes, se produzcan de formas ligeramente diferentes.
Una cadena nueva, que corre de 5' a 3' hacia la horquilla de replicación, es fácil. Esta cadena se produce continuamente porque el ADN polimerasa se mueve en la misma dirección que la horquilla de replicación. Esta cadena sintetizada continuamente se llama cadena líder.
La otra cadena nueva, se aleja de la horquilla, por lo que es más difícil. Esta cadena se produce en fragmentos porque, conforme avanza la horquilla, el ADN polimerasa (que se aleja de la horquilla) debe separarse y volver a unirse al ADN recién expuesto. Esta cadena más difícil, que se produce en fragmentos, se llama cadena rezagada. Los pequeños fragmentos se llaman fragmentos de Okazaki, en honor al científico japonés que los descubrió. La cadena líder puede extenderse a partir de un solo cebador, mientras que la cadena rezagada necesita un cebador nuevo para cada uno de los fragmentos cortos de Okazaki.
Además de las principales proteínas mencionadas anteriormente, se necesitan algunas otras proteínas y enzimas para mantener la replicación del ADN funcionando sin problemas. Una es una proteína llamada pinza deslizante, que mantiene a las moléculas de ADN polimerasa en su lugar al sintetizar ADN. La pinza deslizante es una proteína en forma de anillo e impide que el ADN polimerasa de la cadena rezagada se vaya flotando cuando vuelve a comenzar en un nuevo fragmento Okazaki.
La topoisomerasa también juega un papel importante de mantenimiento durante la replicación del ADN. Esta enzima impide que la doble hélice de ADN que está por delante de la horquilla de replicación se enrolle demasiado cuando se abre el ADN. Actúa haciendo mellas temporales en la hélice para liberar la tensión, las cuales vuelve a sellar para evitar daños permanentes.
Por último, se debe hacer un poco de trabajo de limpieza si queremos que el ADN no contenga ARN ni brechas. El ADN polimerasa, la otra polimerasa que participa en la replicación, elimina los cebadores de ARN y los sustituye por ADN. La enzima ADN ligasa sella las brechas que permanecen después de reemplazar los cebadores.
Modelos de replicación propuestos
Modelo Semiconservativo
Cuando Watson y Crick (1953) propusieron el modelo de la Doble Hélice indicaron que dicho modelo sugería una forma sencilla de replicación. El modelo de replicación propuesto por Watson y Crick suponía que el ADN doble hélice separa sus dos hebras y cada una sirve de molde para sintetizar una nueva hebra siguiendo las reglas de complementariedad de las bases nitrogenadas. Dicho modelo recibió el nombre de Semiconservativo, ya que las dos dobles hélices recién sintetizadas poseen una hebra vieja (una mitad vieja) y otra hebra nueva (mitad nueva).
Frente al modelo Semiconservativo propuesto por Watson y Crick (1953) se postularon otros posibles modelos de replicación del ADN, uno de ellos se denominó Modelo Conservativo y otro Modelo Dispersivo.
Modelo Conservativo: Cuando el ADN doble hélice se replica se producen dos dobles hélices, unas de ellas tienen las dos hebras viejas (intacta, se conserva) y la otra doble hélice posee ambas hebras de nueva síntesis.

Modelo Dispersivo: Cuando el ADN, doble hélice se replica se originan dos dobles hélices, cada una de ellas con hebras que poseen tramos viejos y tramos de nueva síntesis en diferentes proporciones.
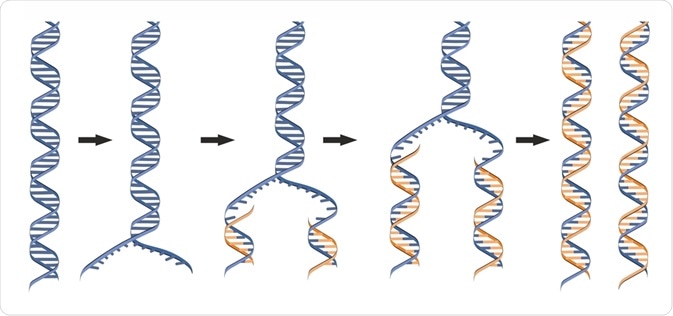
Modelo básico de Watson y Crick sobre la replicación del ADN
Doble hélice de ADN.
Los puentes de hidrógeno se rompen y se abre la hélice.
Cada cadena de ADN actúa como un molde para la síntesis de una nueva cadena complementaria.
La replicación produce dos doble hélices de ADN idénticas, cada una con una cadena nueva y una vieja.
Objetivo
El objetivo de la replicación es el de conservar la información genética. La representación estructural del ADN en doble hélice permite comprender cómo dicha molécula puede dar lugar a otras idénticas, sin perder su conformación. En principio, las dos hebras deberán separarse y, después, mediante la acción de una enzima, añadir desoxirribonucleótidos y, según la complementariedad de bases, construir ADN a partir de las dos hebras molde iniciales.
¿Dónde se lleva acabo?
La replicación se lleva a cabo en la fase de síntesis (S) del ciclo celular. Esta etapa es un paso obligado para realizar la división celular. Por ello, se determina que la información genética se transfiere de una célula a otra mediante el proceso de replicación del ADN. Cada cromosoma es copiado con alta fidelidad en un proceso que involucra un gran número de enzimas. En este proceso la doble cadena de ADN se deshace y cada cadena individual se usa como molde para la producción de la complementaria. El resultado es la producción de dos copias idénticas del material genético.

Traducción del ADN
¿Qué es el ADN?
El ADN es la macromolécula base de la herencia. Es un ácido nucleico que contiene la información de las características hereditarias de cada ser vivo y las secuencias para la creación de aminoácidos que generarán las proteínas vitales para el funcionamiento de los organismos.
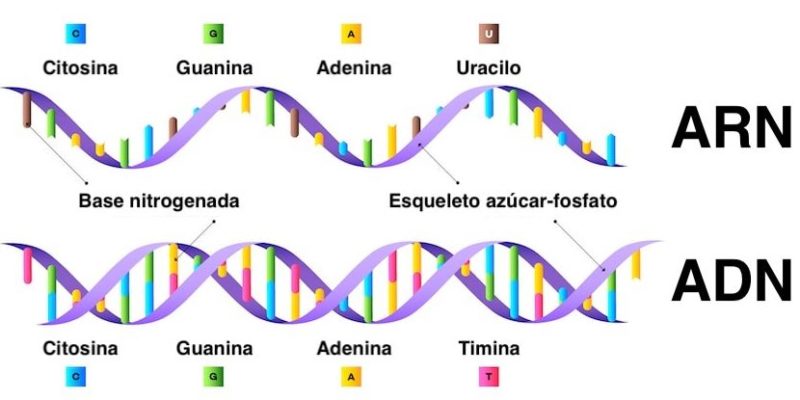
¿Qué es la traducción del ADN?
La traducción es el proceso de traducir la secuencia de una molécula de ARN mensajero (ARNm) a una secuencia de aminoácidos durante síntesis de proteínas. El código genético se describe la relación entre la secuencia de pares de bases en un gen y la secuencia correspondiente de aminoácidos que codifica. En el citoplasma de la célula, el ribosoma lee la secuencia del mRNA en grupos de tres bases para ensamblar la proteína.
¿Qué significa traducción del ADN?
"Traducción" significa literalmente "trasladar", que es lo que significa la traducción. En este caso, lo que se está trasladando es una información que originalmente estaba en el genoma, consagrado en el ADN, para a continuación transcribir a ARN mensajero y luego esa información es traducida del ARN mensajero para una proteína. Teniendo así la misma información, de una forma a otra, de un código de ácidos nucleicos a un código de aminoácidos en una proteína.
Esta traducción no se hace con letras individuales. Es muy parecido al lenguaje humano o cualquier otro idioma en que todas las palabras tienen la misma longitud. Son tres letras, y el lector en este caso se llama ribosoma, que es una gran máquina molecular de subunidades múltiples, que viaja a lo largo del ARNm, y cuando éste detecta cuáles son esas tres letras, decide cual aminoácido debe colocar y es el que se suma a la creciente cadena de aminoácidos, cadena polipeptídica, para convertirse en una proteína.
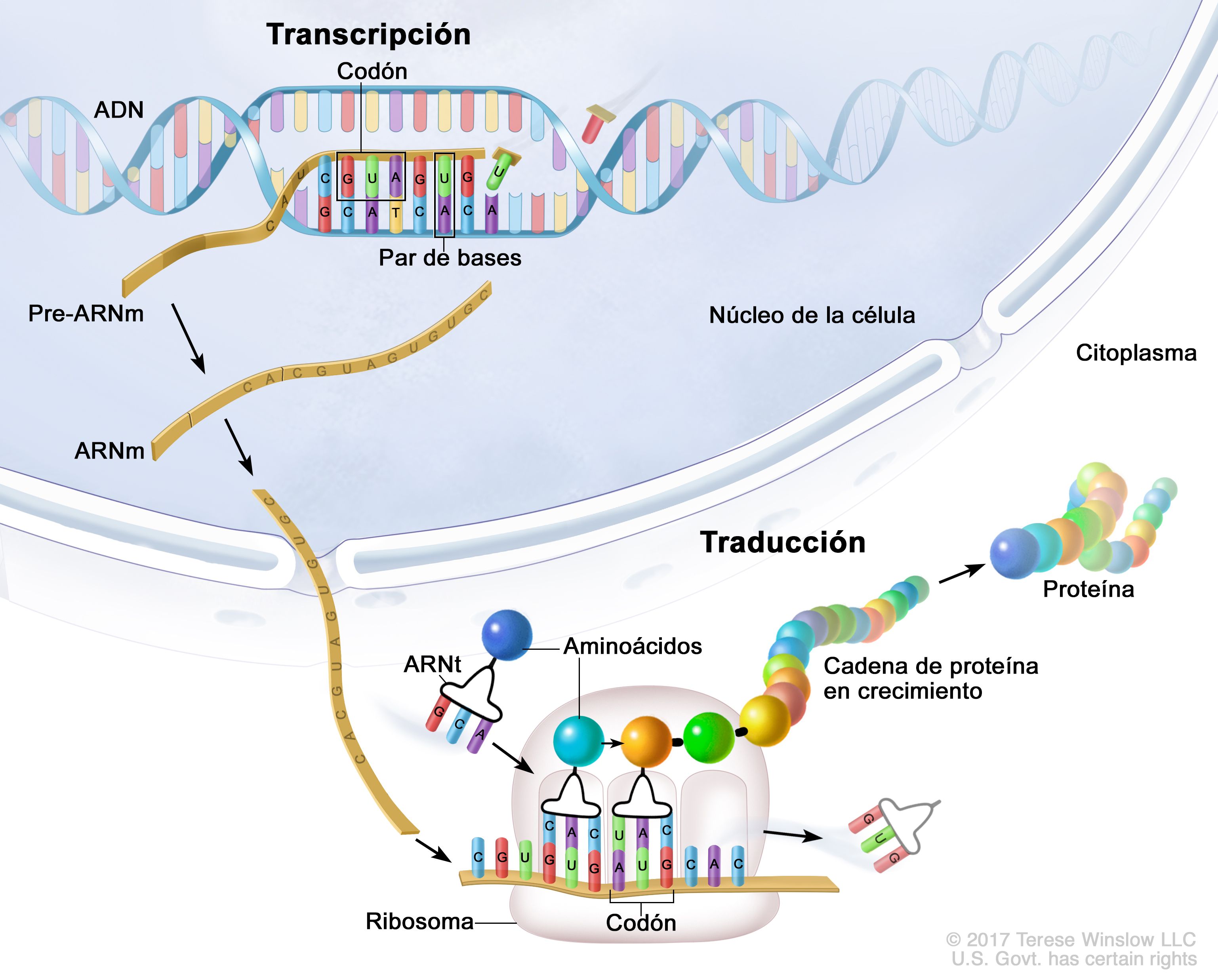
Esas letras del ARNm se llaman un codón, y cada uno de los códigos de un codón codifican para un aminoácido diferente. Y finalmente los aminoácidos son unidos para ensamblar una proteína.

¿Dónde se lleva acabo?
Este proceso ocurre en el citoplasma de la célula y para la mayoría de las proteínas de forma continua, durante todo el ciclo celular a excepción de la etapa M.
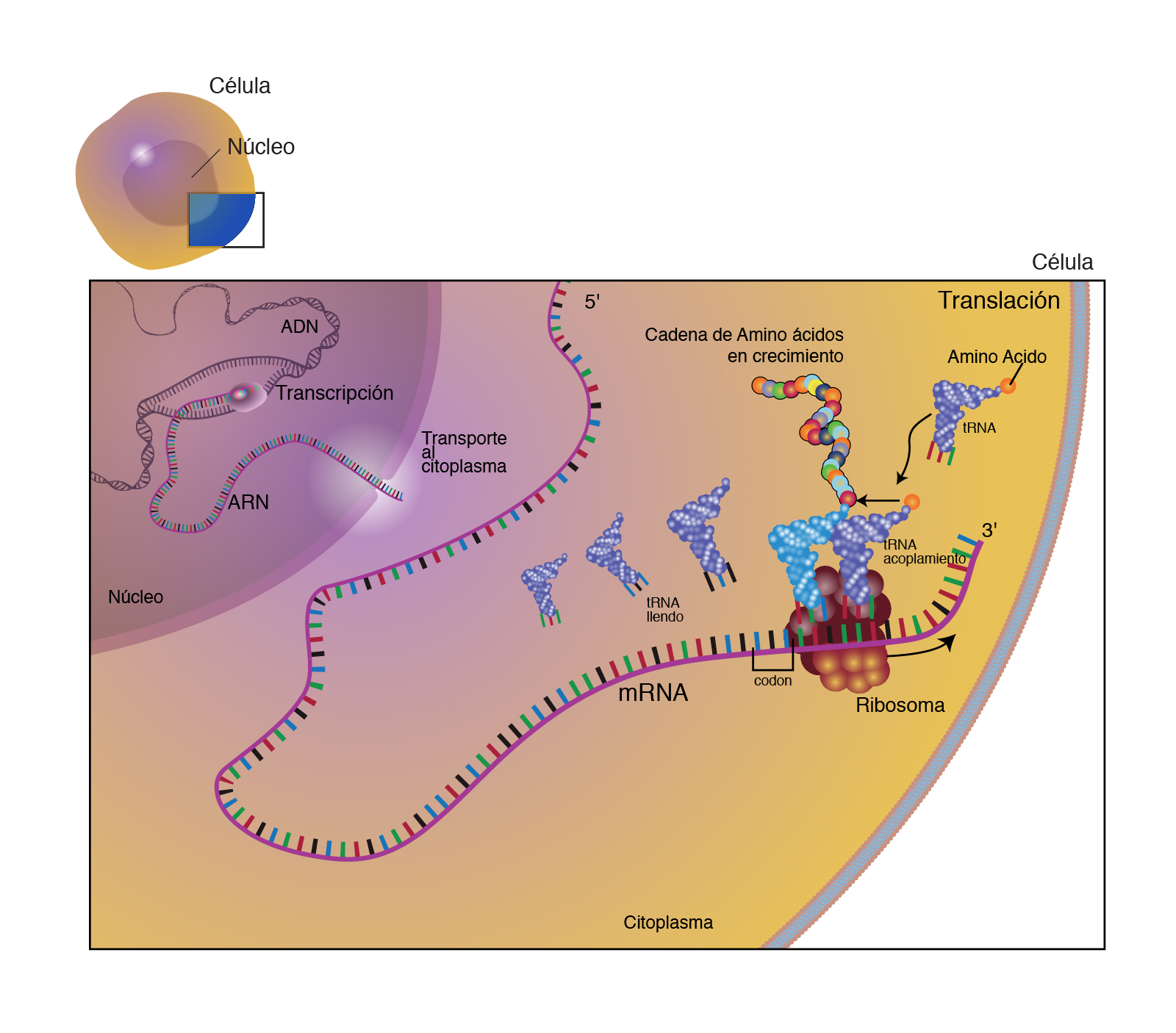
Las funciones de las células siempre van a estar implicadas con las funciones de las proteínas por lo que la expresión de la información genética como proteínas garantiza que las enzimas aceleren las reacciones del metabolismo, los transportadores posibiliten el intercambio de sustancias, los anticuerpos la defensa del organismo, las hormonas proteicas la regulación del metabolismo, los receptores el intercambio de información entre las células, entre otras.
Características
Se caracteriza por ser un proceso gradual y repetitivo, lo que puede ser explicado de la siguiente manera:
Los aminoácidos son añadidos uno a uno por el mismo mecanismo.
La síntesis se realiza de manera unidireccional pues ocurre siempre en dirección N-terminal a C-terminal.
Se realiza de forma colineal a la lectura del ARNm, o sea que la síntesis de la cadena polipeptídica se realiza en la dirección N-terminal a C.terminal, mientras que la lectura del ARNm es en dirección 5'-3'.
Por último, el proceso está acoplado al hidrólisis del GTP: la mayor parte de la energía requerida para el proceso se obtiene del hidrólisis de este nucleótido.
Requerimientos
Es necesario el ARNm, que contiene la información de la proteína que se va a sintetizar.
Proteínas enzimáticas y no enzimáticas, denominadas factores de traducción.
Ribonucleósidos trifosfatados como fuente de energía.
La participación de ARNt que transfiera los aminoácidos al ribosoma.
La presencia de determinados aminoácidos.
Etapas fundamentales
Los especialistas a la hora de estudiar este proceso han elaborado un esquema que contiene cinco etapas básicas, caracterizadas por un conjunto de sucesos y transformaciones únicas para cada una de ellas.
Etapa de Preiniciación
Ocurre la activación de los aminoácidos.
Esta etapa ocurre en el citoplasma y consiste en la unión de cada aminoácido a su ARNt específico.
Esta reacción es catalizada por la enzima aminoacil-ARNt sintetasa. Cada uno de los veinte aminoácidos es reconocido por una aminoacil-ARNt sintetasa específica que pueden presentar de una a cuatro subunidades proteícas. La actividad de ellas es crítica para la exactitud posterior de la traducción pues en el ribosoma ocurre un reconocimiento molecular entre las secuencias del codón y el anticodón, la frecuencia de errores en esta reacción es de 1 en 10000.
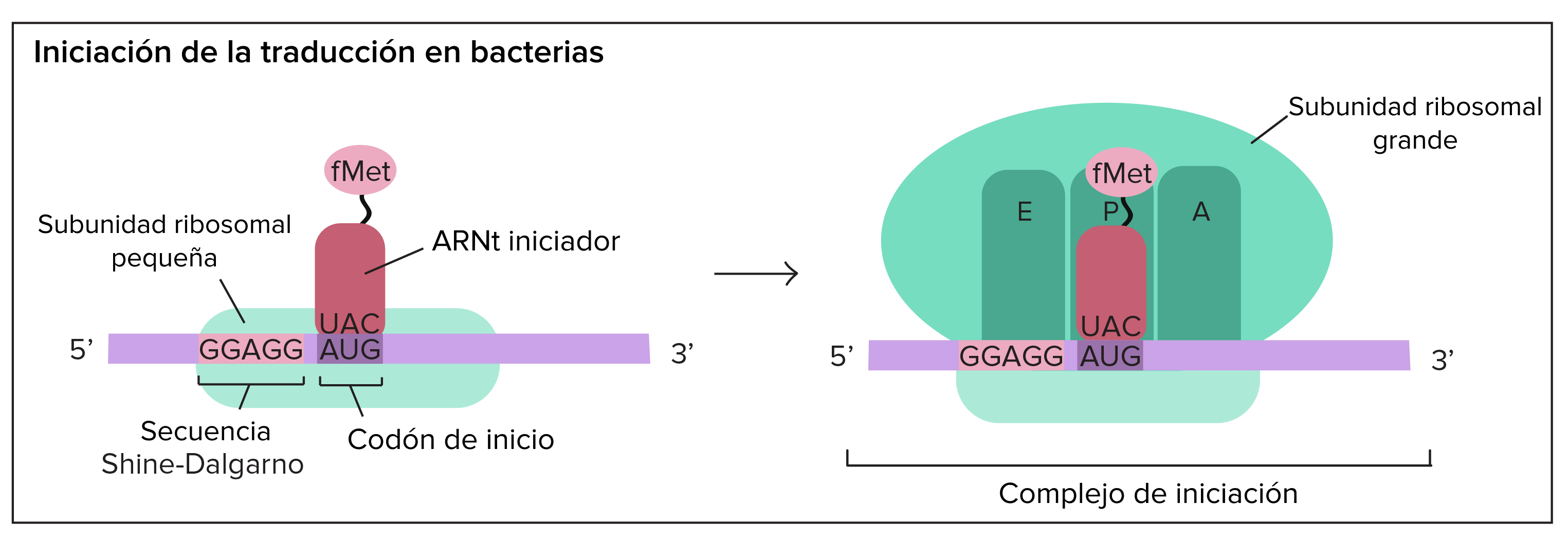
Etapa de Iniciación
El codón de iniciación (AUG) es identificado por el ribosoma con la ayuda de múltiples factores de iniciación (eIF). El codón AUG tiene una doble función: como iniciador, al constituir la señal para el primer aminoacil-ARNt, que es el metionil-ARNt iniciador, y como codón para la incorporación de metionina en el interior de la cadena polipeptídica que crecerá guiada por el ARNm y que corresponde con la etapa de elongación.
La etapa de iniciación ha sido dividida en varias subetapas las que culminan con un ribosoma listo a incorporar al siguiente aminoácido en el sitio A para dar lugar a la siguiente etapa.
Primero ocurre la disociación del ribosoma en sus dos subunidades, lo que es asistido por varios factores de iniciación.
Luego se forma el complejo ternario eIF-2-GTP-Met-ARNtiMet, donde el eIF-2 es una proteína que une al GTP y reconoce al ARNt iniciador (Met-ARNtiMet). Finalmente sucede la unión del complejo ternario a la subunidad menor del ribosoma (40s).
Le sigue el reconocimiento del casquete del extremo 5' del ARNm por el factor de iniciación eIF-4F e incorporación a la subunidad menor.
Como penúltima subetapa se encuentra el movimiento de la subunidad menor unida al ARNm a lo largo del extremo 5', proceso que se denomina scanning y que requiere energía y factores de iniciación adicionales.
Por último, en el momento en que la subunidad menor activada alcanza la posición del codón AUG, la subunidad mayor se une a la menor.

Etapa de Elongación
En esta etapa ocurre la formación del enlace polimerizante durante la formación de la proteína. Aquí, al igual que en las otras etapas, la participación de proteínas adicionales no ribosómicas es fundamental; las que se denominan factores de elongación (eEF). También se divide en fases:
Los aminoácidos activados se unen a un factor de elongación en presencia de GTP. La entrada de cada ARNt al sitio A del ribosoma con un consiguiente gasto de energía (GTP).
El complejo entra al sitio A regido por la complementariedad codón-anticodón por lo que se requiere de una prueba de lectura del codón. Si ocurriera una entrada incorrecta este sería expulsado del sitio para entrar al aa-ARNt correcto.
Cuando los ARNt están correctamente situados ocurre la formación del enlace peptídico con la acción de la peptidil transferasa. En esta fase se localiza el codón que ocupará el sitio A para que pueda entrar el aminoacil-ARNt a dicho sitio. Esto requiere un movimiento del ribosoma denominado translocación y que ocurre simultáneamente con la salida del aminoacil-ARNt descargado. Un factor de elongación (eEF-2) y la energía del GTP son utilizados en esta fase.
Los eventos anteriores se repiten hasta que al sitio A llegue un codón de terminación, que no codifica aminoácido alguno.
Etapa de Terminación
Como los codones de terminación no tienen ARNt que los identifique, en su lugar esto constituye una señal para dar inicio a la terminación de la traducción.
El codón de terminación es reconocido por un factor de liberación unido al GTP. Dicho complejo se une al ribosoma en el sitio A y con la hidrólisis del GTP se produce la liberación de la cadena polipeptídica sintetizada y el desensamblaje de la maquinaria sintetizadora. En estos momentos el ribosoma se encuentra en condiciones de iniciar un nuevo evento de traducción.
Etapa de Posterminación
Esta etapa corresponde a la maduración o procesamiento de la molécula formada.
Durante esta etapa la proteína alcanza su estructura y conformación con su actividad biológica.
Existen diversos eventos que posibilitan que la proteína logre su estado funcional, entre ellos se encuentran: la eliminación de aminoácidos de los extremos y del interior de la cadena; la transformación de los aminoácidos en reacciones de hidrolización, obteniéndose hidroxiprolina y hidroxilisina, aminoácidos que aparecen en el colágeno; incorporación de grupos prostéticos, incorporación de metales en las metaloproteínas, formación de enlaces disulfuro, glicosilaciones y el ensamblaje de subunidades en las proteínas oligoméricas.
Inhibidores
Existen algunos antibióticos que interfieren en el proceso de traducción. Se aprovechan de las diferencias entre los mecanismos de traducción procariota y eucariota para inhibir selectivamente la síntesis de proteínas en las bacterias sin afectar al huésped. Entre ellos se pueden destacar:
La puromicina que tiene una estructura similar al aminoacil-ARNt de la tirosina. Por tanto, se enlaza al sitio A del ribosoma y participa en la formación de enlaces peptídicos, produciendo peptidil-puromicina. Sin embargo, no toma parte en la traslación y se desacopla rápidamente del ribosoma, causando una terminación prematura de la síntesis del polipéptido.
La streptomicina provoca una mala lectura del código genético en las bacterias a concentraciones relativamente bajas e inhibe la iniciación a concentraciones mayores, enlazándose a la subunidad ribosómica 30s.
Aminoglucósidos como la tobramicina y la kanamicina evitan la asociación ribosómica al final de la fase de iniciación y provocan una mala lectura del código genético.
Las tetraciclinas bloquean el sitio A del ribosoma, evitando el acoplamiento de los aminoacil-ARNt.
El cloranfenicol bloquea la fase de la transferencia peptídica de la elongación en la subunidad ribosómica 50s, tanto en las bacterias como en las mitocondrias.
Losmacrólidos y las lincosamidas se enlazan a las subunidades ribosómicas 50s, inhibiendo la reacción de la peptidiltransferasa o la traslación, o ambas cosas.
Localización subcelular
Como localización subcelular de la traducción se pude enunciar a los ribosomas.
Organelos citoplasmáticos no membranosos, integrados por ARNr y proteínas. Los presentes en eucariontes tienen un coeficiente de flotación de 80s, con una subunidad mayor 60s y una menor 40s.

La subunidad mayor presenta los ARN ribosomales: 5s, 5.8s y 28s; a los que se unen aproximadamente 50 tipos diferentes de proteínas. La subunidad menor presenta un solo ARN ribosomal: 18s, al que se unen 30 tipos diferentes de proteínas.
Destacándose en ellos la presencia de tres sitios funcionales importantes:
Sitio A: sitio por donde entran los ARNt con el aminoácido correspondiente al ribosoma.
Sitio P: este es el lugar que ocupa el peptidil-ARNt; tan pronto el ARNt ceda su porción peptidil a la formación del nuevo enlace, pasará al siguiente sitio.
Sitio E: corresponde al sitio ocupado por el ARNt sin el aminoacil ni el peptidil antes de abandonar el ribosoma. Es el sitio de salida de los ARNt descargados.
El código genético

En un ARNm, las instrucciones para construir un polipéptido vienen en grupos de tres nucleótidos llamados codones, algunas características clave de los codones:
Hay 616161 codones distintos para aminoácidos
Tres codones de "alto" indican que el polipéptido ha terminado
Un codón AUG, es la señal de "inicio" para comenzar la traducción (además especifica el aminoácido metionina).
Estas relaciones entre los codones del ARNm y los aminoácidos se conoce como el código genético.
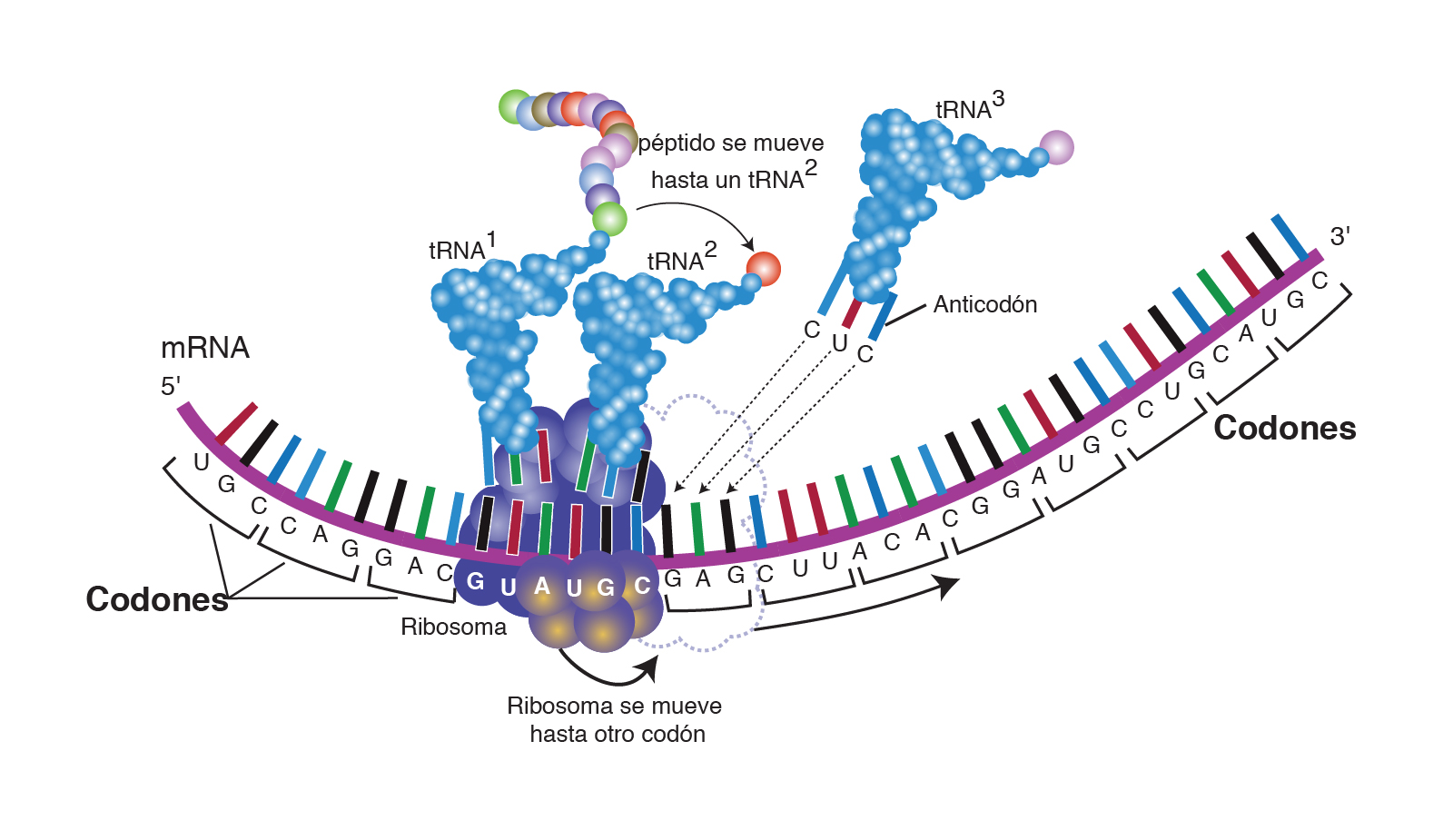
De los codones a los aminoácidos
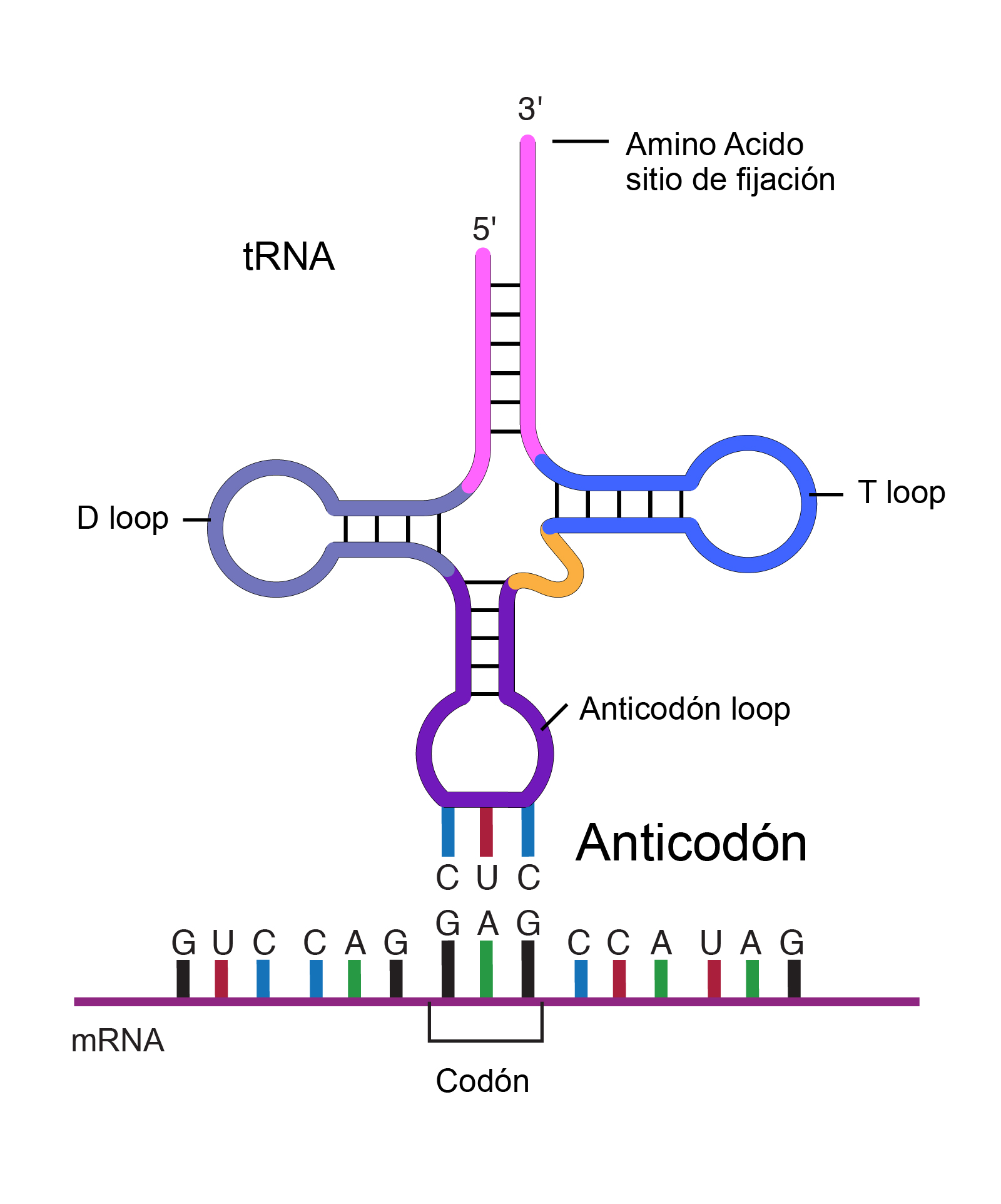
En la traducción, los codones de un ARNm se leen en orden (del extremo 5' al extremo 3') mediante moléculas llamadas ARNs de transferencia o ARNt. Cada ARNt tiene un anticodón, un conjunto de tres nucleótidos que se une a un codón de ARNm correspondiente a través del apareamiento de bases. El otro extremo del ARNt lleva el aminoácido que especifica el codón.
Los ribosomas proporcionan el espacio en el que el ARNm puede interactuar con los ARNt que llevan los aminoácidos.
Hay tres lugares en el ribosoma donde se unen los ARNt: el sitio A, el P y el E. El sitio A acepta un ARNt entrante unido a un aminoácido. El sitio P contiene el ARNt que lleva el polipéptido creciente (el primer aminoácido que se agrega es la metionina (Met)). El sitio E es donde un ARNt se coloca cuando se ha vaciado, es decir, cuando ha transferido su polipéptido a otro ARNt (que ahora ocupa el sitio P).
Los ARNt se unen a los ARNm dentro de una estructura de proteína y ARN llamada ribosoma. A medida que los ARNt entran a los espacios en el ribosoma y se unen a los codones, sus aminoácidos se unen a la cadena de polipéptidos creciente en una reacción química. El resultado final es un polipéptido cuya secuencia de aminoácidos refleja la secuencia de codones en el ARNm.
Importancia
Es el proceso que convierte una secuencia de ARN mensajero en una cadena de aminoácidos para formar una proteína. Es necesario que la traducción venga precedida de un primer proceso de transcripción.
Elementos que intervienen
Los elementos que intervienen en el proceso de traducción son fundamentalmente: los aminoácidos, los ARN-t (ARN transferentes), los ribosomas, ARN-r (ARN ribosómico y proteínas ribosomales), el ARN-m (ARN mensajero), enzimas, factores proteicos y nucleótidos trifosfato (ATP, GTP).
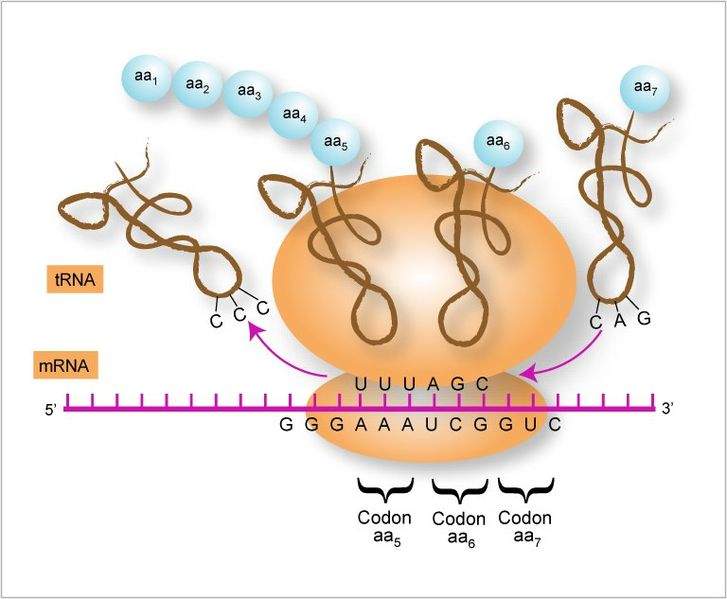
El primer paso que tiene que producirse es la activación de los aminoácidos y formación de los complejos de transferencia. Los aminoácidos por sí solos no son capaces de reconocer los tripletes del ARN-m de manera que necesitan unirse a un ARN de pequeño tamaño (constante de sedimentación 4S) llamado ARN adaptador, ARN soluble o ARN transferente. Crick (1958) postuló la necesidad de la existencia de un adaptador que acoplará cada aminoácido a su correspondiente codón.
Estructura de los ARN Transparentes (ARN-t)
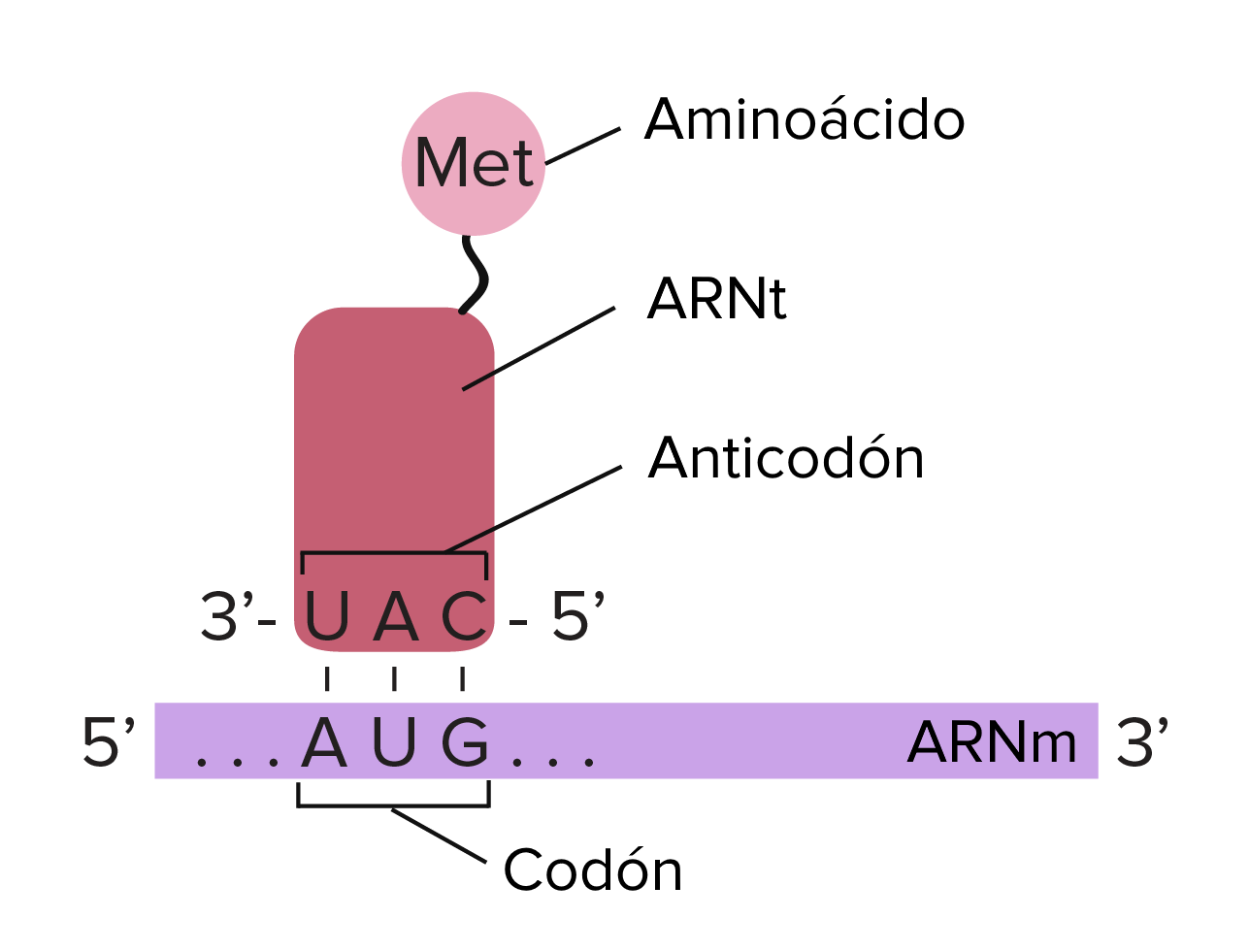
Las moléculas encargadas de transportar los aminoácidos hasta el ribosoma y de reconocer los codones del ARN mensajero durante el proceso de traducción son los ARN transferentes (ARNt). Los ARN-t tienen una estructura en forma de hoja de trébol con varios sitios funcionales:
Extremo 3': lugar de unión al aminoácido (contiene siempre la secuencia ACC).
Lazo dihidrouracilo (DHU): lugar de unión a la aminoacil ARN-t sintetasa o enzimas encargadas de unir un aminoácido a su correspondiente ARN-t.
Lazo de T ψ C: lugar de enlace al ribosoma.
Lazo del anticodón: lugar de reconocimiento de los codones del mensajero
Normalmente el ARN-t adopta una estructura de hoja de trébol plegada en forma de L o forma de boomerang.
Bibliografía
Significado de ADN (ácido desoxirribonucleico) (Qué es, Concepto ...
Traducción | NHGRI
Traducción del ADN – EcuRed
¿Cuál es la importancia de la traducción del ADN? - Brainly.lat
PROCESOS GENÉTICOS DE LA SÍNTESIS DE PROTEÍNAS ...
Transcripción del ADN
¿Qué es el ADN?
El ácido desoxirribonucleico (ADN) es un ácido nucleico que contiene toda la información genética hereditaria que sirve de “manual de instrucción” para desarrollarnos, vivir y reproducirnos.
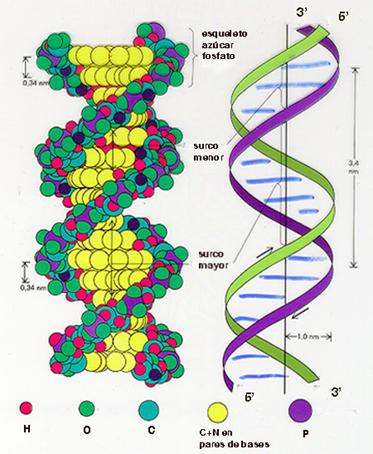
El ADN se encuentra en el núcleo de las células, aunque una pequeña parte también se localiza en las mitocondrias, de ahí los términos ADN mitocondrial y ADN nuclear. El ADN como ácido nucleico está compuesto por estructuras más simples, las bases nitrogenadas.
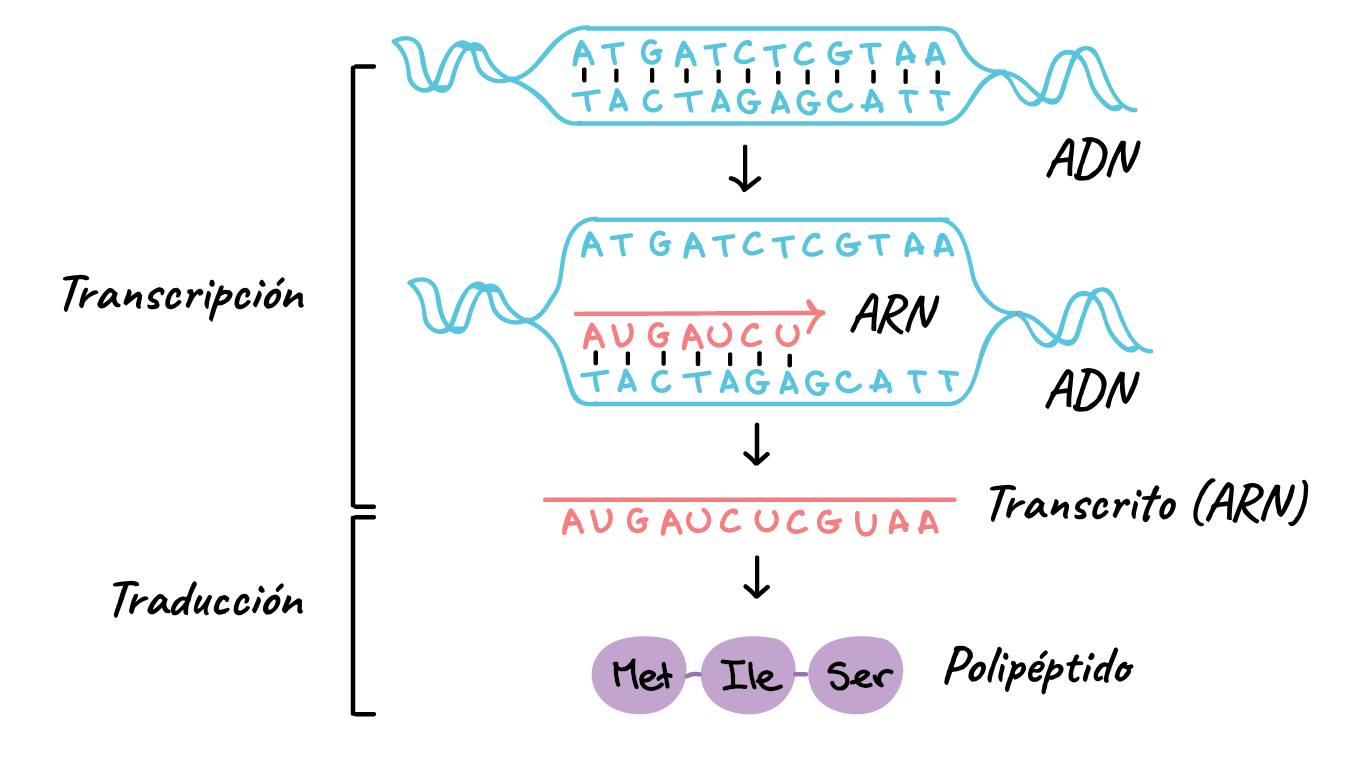
¿Qué es Transcripción?
La transcripción del ADN es el primer proceso de la expresión genética, mediante el cual se transfiere la información contenida en la secuencia del ADN hacia la secuencia de proteína utilizando diversos ARN como intermediarios.
Durante la transcripción genética, las secuencias de ADN son copiadas a ARN mediante una enzima llamada ARN polimerasa (ARNp) la cual sintetiza un ARN mensajero que mantiene la información de la secuencia del ADN.
De esta manera, la transcripción del ADN también podría llamarse síntesis del ARN mensajero. Es un proceso mediante el cual los genes que se encuentran en el ADN de los cromosomas son selectivamente localizados, reconocidos y transcritos, produciendo ARN mensajero, ribosomal (estructural) y de transferencia (adaptadores). Puede ser descrito como mecanismo básico de complementariedad de bases, añadidas en forma gradual, unidireccional y antiparalela, sin necesidad de un iniciador y acoplada al hidrólisis del pirofosfato
Objetivo
El objetivo de la transcripción es producir una copia de ARN de la secuencia de ADN de un gen. En el caso de los genes codificantes, la copia de ARN, o transcrito, contiene la información necesaria para generar un polipéptido (una proteína o la subunidad de una proteína). Los transcritos eucariontes necesitan someterse a algunos pasos de procesamiento antes de traducirse en proteínas.
Características
La síntesis de ARN dependiente de ADN es un proceso muy parecido al de la replicación, existiendo una serie de similitudes que establecen un estrecho modo de operación por parte de la célula a la hora de procesar el material genético. Así puede observarse el hecho de que la reacción es igualmente de polimerización, se necesita también un molde para realizarla, y, por último, la dirección de síntesis es fija al igual que en la replicación.
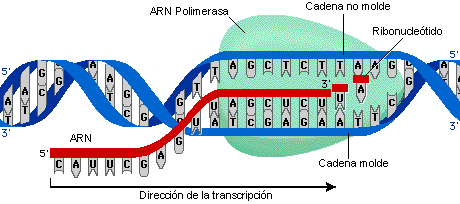
Sin embargo, la transcripción presenta una serie de características que la diferencian de la replicación, como son:
1) El proceso se limita a una porción de ADN, se dice que es un proceso selectivo, ya que ha de reconocerse un punto de inicio y uno de terminación en la molécula de ADN.
2) El proceso puede repetirse infinidad de veces a lo largo de la vida de la célula, a diferencia de la replicación que es un proceso que marca la división celular, se dice que es reiterativo. Una región concreta de ADN puede ser copiada multitud de veces dando lugar a la formación de múltiples moléculas iguales de ARN.
3) El proceso no afecta a la estructura del ADN, es un proceso conservador de la molécula de ADN, el gen o genes copiados permanecen iguales.
4) El proceso es monocatenario, afecta a una sola de las cadenas del ADN, y la copia resultante, o ARN es una molécula de una única cadena o monocatenaria. La situación de los genes a copiar puede localizarse en cualquiera de las dos cadenas del ADN, la cadena que funciona como molde para la síntesis de ARN se la denomina hebra molde (-), y la cadena complementaria hebra no molde (+). Genes diferentes pueden usar diferentes cadenas como molde.
Requerimientos
Para que el proceso de formación de cada uno de los ARN se complete con éxito durante la transcripción es necesario que un exista un gen que codifique al ARN, así como otras secuencias reguladoras de su expresión. También debe existir una ARN polimerasa encargada de la unión de los ribonucleótidos mediante enlaces polimerizantes 3'-5' fosfodiéster.
Es imprescindible la presencia de factores de transcripción (proteínas que facilitan el ensamblaje del complejo de transcripción y la acción de las ARN polimerasas). Sin olvidar a los ribonucleótidos que sirven de sustrato y a las enzimas especiales que se encargan de la maduración o procesamiento de los ARN para que posean toda su funcionabilidad.
Etapas fundamentales

Para el mejor estudio del proceso de transcripción este puede ser dividido en cinco etapas:
Etapa de Preiniciación
Los eventos de Preiniciación consisten en la localización, por parte de la ARN polimerasa, de un sitio específico del ADN y la separación de las dos cadenas, de forma que la secuencia de bases sea accesible a la enzima. Por convención se representa a la hebra de ADN, que no es copiada, y se designa como la hebra codificante por ser muy parecida en su secuencia al ARN, esta se debe escribir de izquierda a derecha en el sentido 5'-3'. La otra hebra se denomina molde porque esta es precisamente su función.
La primera base que se transcribe se toma como referencia y se designa como +1. Las escritas a su izquierda llevan números negativos y las que van hacia la derecha, números positivos, la posición 0 no existe.
El elemento central de la regulación de la transcripción es el promotor, que se define como un segmento de ADN; este contiene las señales para la unión adecuada de la holoenzima de la ARN polimerasa y su posterior activación para formar un sitio capaz de iniciar la transcripción. El promotor contiene, además, en sí o en sus alrededores, otras señales que controlan la unión específica de las proteínas reguladoras; estas proteínas modulan la efectividad de la unión de la polimerasa con el promotor.
El reconocimiento entre la ARN polimerasa y el promotor se debe a la presencia de un grupo de donantes y aceptores de puentes de hidrógeno orientados específicamente, que interactúan con grupos similares en el dominio de unión de la enzima; estos determinan la especificidad del reconocimiento.
A la estabilidad contribuyen otras interacciones menos específicas, pero más fuertes, con las interacciones iónicas entre las cargas negativas de la parte monótona de la estructura del ADN y residuos básico del dominio de unión de la proteína, y quizás interacciones hidrofóbicas entre el metilo de la timina y grupos apolares de la superficie de la enzima.
La unión de la polimerasa en sitio adecuado provoca un desenrollamiento local del ADN (favorecido por el elevado contenido de pares AT) que incluye aproximadamente las bases de -10 a +5. Esta estructura es muy estable y recibe el nombre de "promotor abierto". Una vez formado el promotor abierto, la enzima comienza a deslizarse sobre el ADN, hasta arribar a la primera base que va a ser copiada.
Etapa de Iniciación
La iniciación de la transcripción comienza con la unión al complejo, formado por la polimerasa y el promotor abierto del ribonucleósido trifosfatado que formará al extremo 5'-P de la cadena naciente de ARN.
La unión del segundo nucleótido y la formación del enlace fosfodiéster forma también parte de la iniciación; por lo tanto, consiste en la formación de un complejo ternario entre la polimerasa, el ADN y un segmento de ARN, lo suficientemente largo para que el complejo sea estable y no se disocie.
La ARN polimerasa contiene dos sitios de unión para nucleósidos trifosfatados, llamados sitios de iniciación y de elongación. El primero une frecuentemente nucleótidos de purina, casi siempre ATP, luego la primera base en ser copiada es casi siempre la timina. Una vez formado par AT se incorpora otro nucleósido trifosfatado al sitio de elongación; solo se incorporará aquel capaz de formar un par con la base +2. Cuando los dos sitios están ocupados se produce la reacción y queda formado el primer enlace fosfodiéster.
Inmediatamente después se produce el movimiento de la polimerasa hacia el nucleótido siguiente. El final de la iniciación está determinado por la separación del factor σ y la formación de un complejo ternario estable.
Etapa de Elongación
Después de ser liberado el factor σ, la polimerasa adquiere una nueva conformación y se forma un complejo ternario (polimerasa-ADN-ARN naciente) que se caracteriza por su alta estabilidad. En esta etapa se pueden describir los siguientes pasos:
Unión a la enzima del nucleósido trifosfatado sustrato de forma específica para la ribosa y la base.
Ataque nucleofílico del P(α) al 3'-OH con la consiguiente formación del enlace fosfodiéster y la generación de pirofosfato.
La polimerasa es traslocada a lo largo de la cadena de ADN produciéndose un desenrollamiento por delante y un enrollamiento por detrás.
La zona de elongación tiene una longitud constante de 17pb, se supone que esto se deba a una propiedad de la enzima y no a la secuencia de bases en la cadena de ADN. Un detalle importante es que el desplazamiento de la polimerasa no se produce a una velocidad constante; hay zonas en que le desplazamiento es más rápido que en otras, incluso existen pausas donde la enzima se detiene totalmente, estas pausas están relacionadas con las zonas ricas en pares CG. La polimerasa en su movimiento va desenrollando la doble hebra de ADN y esto es más difícil donde predominan los pares CG.
Etapa de Terminación
La terminación de la síntesis del ARN ocurre en sitios de secuencias de bases específicas en la molécula de ADN. Todo parece indicar que la formación de la estructura en "tallo y asa", en la molécula de ARN, está implicada notablemente en el mecanismo de la terminación. Se ha comprobado que mientras mayor sea el contenido de pares CG de dicha estructura y mayor la longitud de la cola de poli(U), la terminación será más efectiva.
No se conoce con exactitud cómo se produce la terminación, pero parece estar relacionada con una pausa en el movimiento de la polimerasa. El proceso incluye: el cese de la elongación de la cadena de ARN, la liberación del ADN neoformado y la liberación de la ARN polimerasa del ADN.
Etapa de Posterminación
Acerca del proceso de posterminación o maduración de los ARN en procariontes se pueden hacer cuatro afirmaciones o valoraciones basadas en los resultados obtenidos de experimentos prácticos realizados fundamentalmente:
Todos los ARNt y los ARNr son procesados exceptuándose de este proceso los ARNm.
Para generar el extremo 3'-OH y el 5'-P se requieren cerca de diez tipos de ARNasa
Durante el evento de reconocimiento las ARNasas favorecen a las estructuras tridimensionales antes que a las secuencias de bases.
Se forman bases raras por transformación enzimática de las bases comunes.

La ARN polimerasa
La principal enzima que participa en la transcripción es la ARN polimerasa, la cual utiliza un molde de ADN de cadena sencilla para sintetizar una cadena complementaria de ARN. Específicamente, la ARN polimerasa produce una cadena de ARN en dirección de 5' a 3', al agregar cada nuevo nucleótido al extremo 3' de la cadena.
Inhibidores de la transcripción
Pueden ser considerados dos tipos fundamentales de inhibidores de este proceso: los que impiden la separación de las cadenas de ADN, que por lo general son también inhibidores de la replicación, y los que actúan de forma directa sobre las polimerasas. Entre ellos se encuentra el antibiótico rifampicina, que actúa sobre la ARN polimerasa impidiendo la fase de iniciación.
Una sustancia denominada α-amanitina que inhibe la síntesis de ARm en organismos eucariotas pues actúa sobre la ARN polimerasa. Estos inhibidores presentan importantes aplicaciones terapéuticas y un inapreciable valor en el uso de laboratorio.
Enzimas que participan en la Síntesis de ARN
A nivel de las células procariotas, la enzima que se describió en primer lugar fue una ARN polimerasa dependiente de ADN, encargada de formar los distintos tipos de ARN a partir de los ribonucleótidos activados (ATP, GTP, CTP y UTP). La forma de establecer el enlace fosfodiéster consiste en unir el nucleótido entrante por su extremo 5' a la cadena en crecimiento por su extremo 3' libre, siendo por lo tanto la dirección de síntesis 5'→3'. Para la síntesis sólo se usa una cadena de ADN molde que es copiada en su dirección 3'→5', la cadena de ARN en formación queda situada en dirección antiparalela a la cadena molde de ADN.
Cada uno de los nucleótidos de la cadena en formación, guarda el principio de complementariedad de bases con la salvedad de que las bases de adenina del ADN copiado darán lugar a bases de uracilo en la copia de ARN.

Una diferencia importante de esta enzima es que no requiere cebador pudiendo comenzar la síntesis con los dos ribonucleótidos iniciales. El punto de inicio viene “señalado” en el ADN por unas secuencias concretas denominadas promotores, siendo característico que el primer ribonucleótido sea púrico y conserve todos sus grupos fosfato.
A lo largo del proceso se forma una doble hélice entre el ADN y el ARN en formación, que recibe el nombre de híbrido ADN-ARN.
La ARN polimerasa es una enzima compleja formada por 5 subunidades (α α β β’ ω) que forman su núcleo y una sexta subunidad (σ), que se encuentra unida a la enzima en los momentos iniciales del proceso. Carece de actividad exonucleasa lo cual implica que, en el proceso de polimerización, que desarrolla a una velocidad media de unos 50 nucleótidos por segundo (entre 30 y 85), se cometan errores en una proporción de uno por cada 104 o 105 ribonucleótidos incorporados.
Maduración del ARN
La mayor parte de las moléculas de ARN procariotas y la totalidad de las eucariotas recién sintetizadas, los denominados transcritos primarios, han de pasar por una serie de modificaciones o cambios que se conocen con el nombre de maduración del ARN o procesos postranscripcionales.
Una de las características más sorprendentes que se producen en este proceso, es la participación de moléculas de ARN que tienen actividad catalítica o enzimática. Los transcritos primarios de los ARNm y ARNt son los que experimentan más modificaciones.
Dentro de todos los posibles sistemas de maduración se analizan cuatro tipos fundamentales.
1) Corte y empalme.
En el caso de los ARNm de eucariotas que llevan información de ungen (monocistrónicos), las secuencias con información para el polipéptido no están contiguas, sino que están separadas por segmentos de ARN sin función codificante.
Estas secuencias no codificantes se denominan intrones, y las secuencias codificantes, exones. Para obtener un ARNm funcional se han de eliminar los intrones a través de un proceso denominado de corte y empalme (“splicing”), permitiendo que los exones formen una secuencia ininterrumpida.
Existen varios tipos de intrones que se diferencian por su forma de ser eliminados de la secuencia polinucleotídica. Algunos experimentan autocorte y empalme, sin necesidad de participación de proteínas; otros, los mayoritarios, requieren la acción de un complejo ARN-proteína para realizar la reacción de corte y empalme.
El ARN que participa en este complejo es el nuclear pequeño (ARNsn), con cinco tipos moleculares, que forma complejos con proteínas denominados “ribonucleoproteínas”.
2) Corte.
Los ARN ribosómicos, tanto de procariotas como de eucariotas, son sintetizados como largos transcritos primarios, que darán origen mediante secciones o cortes adecuados a los distintos tipos moleculares de ARNr. Los ARNt, con 40 ó 50 tipos diferentes por célula, se forman a partir de transcritos más grandes, que posteriormente son cortados por sus extremos 3' ó 5'.
3) Modificaciones de adición.
Los ARNm de células eucariotas se caracterizan por presentar rasgos comunes en ambos extremos de la cadena. La mayoría tiene en su extremo 5' un casquete formado por un nucleótido mutilado de guanosina unido a través de un enlace 5'-5'trifosfato. En el extremo3' tiene una cola de poliA formada por 20 a 250 residuos adenilato.
Los ARNt también presentan modificaciones en los extremos, en el 3' es añadida la secuencia nucleotídica CCA, catalizada por la enzima ARNnucleotidiltransferasa.
4) Modificación de bases.
En los ARNt, existen por último modificaciones químicas realizadas sobre las bases, como metilaciones, desaminaciones o reducciones; estas bases situadas en lugares concretos de la estructura del ARNt determinan su estructura espacial o conformación natural.
Bibliografía
ADN y ARN concepto, diferencias y funciones | VIU
Transcripción genética - Wikipedia, la enciclopedia libre
Transcripción del ADN – EcuRed
Resumen de la transcripción (artículo) | Khan Academy
Tema 7C - Bloque I: Transcripción