subgrupo de antimicrobianos con actividad antibacteriana.
Clasificación
Origen
Semisintéticos
Modificaciones químicas de moléculas sintetizadas por organismos vivos. Como ampicilina.
Naturales (biológicos)
Sintetizados por organismos vivos. Penicilina, cloranfenicol.
Sintéticos
Generados mediante síntesis química.
Actividad microbiana
Matan al microorganismo por lisis. Penicilina
• Bactericidas
• Bacteriostáticos
• Bacteriolíticos
Microorganismos susceptibles.
• Antiprotozoarios: Meticilina
• Antibacterianos
• Antivíricos: Foscarnet
• Antifúngicos: Amfotericina B
Estructura química
Betalactámicos
Cefalosporinas
• 1ª Generación
• 2ª Generación: Cefaclor, Cefalexina, Cefalexina.
• 3ª Generación: Cefazolina, Ceftriaxona, Ceftazidima, Cefixima.
• 4ª Generación: Cefepime.
• Monobactámicos: Aztreonam
• Carbapenemicos: Meropenem, Doripenem, Ertapenem.
Quinolonas
• 1ª Generación: Ácido pipemídico
• 2ª Generación: Norfloxacino, Ciprofloxacino.
• 3ª Generación: Levofloxacino.
• 4ª Generación: Moxifloxacino, Nadifloxacino.
Aminoglucósidos: Gentamicina, Amikacina, Neomicina, Tobramicina
Tetraciclinas: Doxiciclina, Minociclina.
Macrolidos: Claritromicina, Azitromicina, Eritromicina.
Lincosamidas.
Espectro de acción
Intermedio
Cubren varios tipos de microorganismos pero no todas las especies de cada grupo.
Amplio
Estrecho
Tienen actividad microbiana sobre un grupo reducido de especies.
Mecanismos de acción y resistencia
1. Inhibidores de la formación de la pared bacteriana
2. Inhibidores de la síntesis proteica
3. Inhibidores de la duplicación del ADN
4. Inhibidores de la membrana cit.
5. Inhibidores de vías metabólicas.
Betalactámicos.
Inhibidores de la síntesis de la pared bacteriana.
• Efecto autolítico.
• Inhibición de la última parte de la síntesis de peptidoglicano.
• Necesaria fase de multiplicación (síntesis de pared celular).
• Acción de las PBP.
Mecanismo de resistencia.
• Producción de betalactamasas: enzimas que hidrolizan anillo beta lactámico.
Penicilinas. Cefalosporinas. Monobactámicos. Carbapenems.
Glicopéptidos.
Mecanismos de acción.
• Los glicopéptidos inhiben la síntesis y el ensamblado de la segunda etapa del peptidoglicano de la pared celular.
• Alteran la permeabilidad de la membrana citoplasmática.
• Altera la síntesis de ARN.
• Sus múltiples mecanismos de acción contribuyen a la baja frecuencia de desarrollo de resistencia. Se une rápida y firmemente a las bacterias y ejerce su efecto bactericida.
Mecanismo de resistencia: Mecanismo inducible, que requiere la presencia de este antibiótico. El mecanismo se pone en funcionamiento mediante un sistema de traducción de señales
de dos componentes: un péptido transmembrana denominado vanS y un segundo mensajero llamado vanR.

Aminoglucósidos.
Mecanismo de acción.
• Se unen de forma irreversible a la subunidad 30S del ribosoma, consiguiente bloqueo de la síntesis proteica de la bacteria.
• Bactericidas de acción rápida (dpte CIM).
• Tienen EPA.
• Deben ingresar ala célula para actuar: entran a bact G - por porinas (limitantes: disminución del PH, anaerobiosis), incorporan aminoglucósidos en el interior de la bacteria, especialmente en los cocos grampositivos.
Mecanismos de resistencia: Inactivación enzimática, seguido por alteración de la permeabilidad, limitado a la estreptomicina y la espectinomicina, puede observarse una mutación puntual en el sitio de acción de estos agentes, la proteína de la subunidad 30s denominada proteína S12.

Macrólidos.
Mecanismo de acción: Se unen a la subunidad 50S del ARN ribosómico en forma reversible.
El mecanismo de resistencia de eflujo activo ya ha sido mencionado y es mediado por plásmidos. Dos tipos de alteraciones del sitio blanco pueden producir resistencia a macrólidos
Quinolonas.
Mecanismo de acción: Actúan inhibiendo la ADN girasa, enzima que cataliza el superenrollamiento del ADN cromosómico, que asegura una adecuada división celular.
La resistencia a quinolonas en bacterias gramnegativas está causada fundamentalmente por mutaciones cromosómicas.
Mecanismos de acción.
Mecanismo de resistencia antimicrobiana (AMR)
Fenómeno natural por el cual microorganismos ganan resistencia a los efectos de los fármacos antimicrobianos, como los antibióticos, que anteriormente eran eficaces en el tratamiento de infecciones.
La resistencia natural es propia de cada familia, especie o grupo bacteriano.
La resistencia adquirida es variable y es adquirida por una cepa de una especie bacteriana.
Genética de la resistencia
Mutaciones cromosómicas
En este caso se producen cambios en la secuencia genómica que posibilitan la resistencia.
Se trata de mutaciones que presentan una transmisión vertical (se transmiten a la descendencia), aparecen de forma espontánea, son irreversibles y aparecen tras la administración del antibiótico.
Resistencia extracromosómica
En este tipo de resistencia adquirida se produce la transmisión de material genético extra cromosómico como plásmidos, transposones e integrones.
Los plásmidos que transportan los genes de resistencia a los antibióticos se denominan plásmidos R.
Inactivación enzimática
El principal mecanismo de inactivación es la hidrólisis, como sucede con las betalactamasas y los betalactámicos.
Disminución de la penetración del antibiótico al lugar de acción.
La bacteria se hace resistente al reducir la cantidad de antibiótico que penetra en el medio intracelular.
Este fenómeno es debido a una disminución de la permeabilidad (fosfomicina, ampicilina) o a la presencia de una bomba de expulsión activa (tetraciclina).
Modificación del lugar de acción del antibiótico.
Cambios estructurales en los lugares de acción ocasionan una menor afinidad por el antibiótico lo que genera una disminución de su eficacia clínica.
Interacciones entre fármacos antibacterianos.
Este fenómeno se puede dar al combinar un antibiótico bactericida con otro bacteriostático (betalactámicos junto con tetraciclinas) o al administrar dos antibióticos que actúen en el mismo lugar de acción, ya que compiten por la diana (cloranfenicol y macrólidos).
Mecanismos de resistencia a los antibióticos que actúan sobre la pared bacteriana.
Peptidoglicano, actúa como una armadura o malla envolviendo la bacteria, ofreciéndole rigidez y estabilidad.
Un plásmido es una pequeña molécula de ADN circular que a menudo se encuentran en bacterias y otras células. Los plásmidos son separados del cromosoma bacteriano y se replican independientemente de ella.
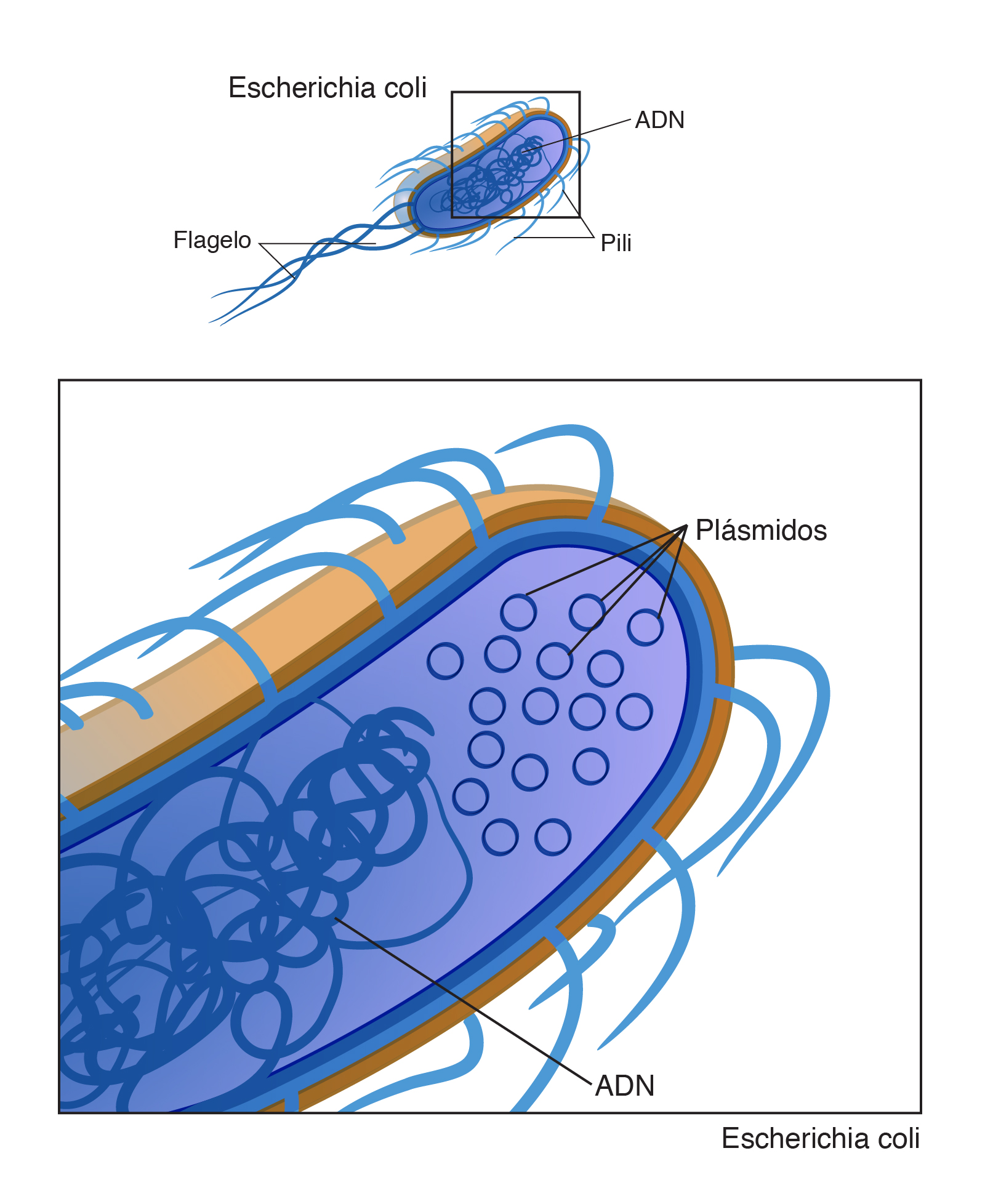
En 1952 Lederberg propuso cambiar el término de episoma a plásmido, limitando el uso para elementos extracromosomales que se integran al cromosoma. Se reconoció a partir de esto, que los plásmidos juegan un papel importante en la evolución bacteriana, en especial en transferencia de resistencia a antibióticos y patogenicidad.

Esther Lederberg.
Antibióticos.
Técnicas para identificación de resistencia a antibióticos
Técnicas moleculares
Permiten la detección de material genético, tanto ácido desoxirribonucleico (ADN) como ácido ribonucleico (ARN).
Microarrays
Se basa en la detección, mediante un análisis de imágenes, de la hibridación de una molécula diana a una sonda específica inmovilizada en un soporte sólido.
Métodos comerciales de antibiograma
Las tiras comerciales con un gradiente de antibiótico se han utilizado para realizar un antibiograma directo a partir de muestras respiratorias. La muestra se siembra en masiva en placas de agar Mueller Hinton y seguidamente se depositan las tiras con antibiótico.
Técnicas inmunocromatográficas
En el caso de resistencias a antibióticos permiten detectar enzimas bacterianas que hidrolizan el antibiótico. El procedimiento consiste en suspender la bacteria en un diluyente.
Métodos colorimétricos
En estos test, la bacteria se incuba en presencia del antibiótico.
Métodos de imagen
Se monitoriza, mediante la toma de imágenes, el crecimiento bacteriano de una cepa incubada en presencia de diferentes concentraciones de antibiótico.
Nefelometría
Mide la intensidad de radiación dispersa que se genera cuando un haz de luz atraviesa una suspensión de partículas.
Espectrometría de masas MALDI-TOF
Predice en menos de 3h si las bacterias producen enzimas que hidrolizan los antibióticos. Se realiza una centrifugación y el sobrenadante obtenido se analiza mediante MALDI-TOF
Citometría de flujo
Técnica basada en la formación de un flujo de partículas (generalmente células) ordenadas en fila con una intensidad de 500-4.000 partículas/segundo.
Quimioluminiscencia y bioluminiscencia
Emisión de luz que ocurre en ciertas reacciones químicas cuando las moléculas excitadas vuelven al estado fundamental. Para la obtención de luz solo se requiere la adición de menadiona al cultivo del microorganismo.
Microfluidos
S realice en plataformas o chips que llevan a cabo, utilizando pequeños volúmenes de trabajo, múltiples ensayos que aportan información acerca de la sensibilidad de las bacterias a los antibióticos,
Métodos de lisis bacteriana
La bacteria es incubada en presencia del antibiótico a la concentración deseada; seguidamente la bacteria se inmoviliza en un microgel de agarosa y es expuesta a una solución de lisis que produce la liberación del ADN.